Le tecnologie di intelligenza artificiale si stanno rivelando strumenti preziosi per aumentare l’efficienza nella scoperta di nuovi antibiotici contro batteri multi-resistenti, imminente minaccia per la salute pubblica. Questi sistemi computazionali permettono di processare enormi quantità di dati, tagliando tempi e costi nel processo di drug discovery, ma sono ancora considerati delle “black boxes” dal funzionamento oscuro.
In uno studio pubblicato a fine anno sulla rivista scientifica Nature, i ricercatori del Massachusetts Institute of Technology (MIT, USA) hanno aperto la black box dell’algoritmo di deep learning con il quale hanno identificato una nuova classe di antibiotici contro un ceppo resistente di Stafilococcus aureus.
Nuova classe di farmaci dall’intelligenza artificiale
Come già riportato su Agenda17, per contrastare i batteri che diventano resistenti agli antibiotici attuali, gli sforzi della ricerca si stanno ora concentrando nell’individuare nuove classi di farmaci, sufficientemente diverse da quelle già esistenti. La nuova classe di antibiotici identificata recentemente dai ricercatori americani è in grado di agire specificamente contro le membrane batteriche di Stafilococcus aureus – meticillina resistente (MRSA), un batterio responsabile di infezioni a pelle, polmoni, valvole cardiache e ossa, recentemente diventato resistente a trattamenti con diversi antibiotici.
L’approccio utilizzato dai ricercatori integra dati sperimentali e analisi computazionali con strumenti di deep learning, una branca del machine learning. La tecnologia alla base di questi algoritmi è costituita da reti neurali artificiali, composte da nodi interconnessi tra loro, come i neuroni che costituiscono il nostro sistema nervoso. “Hanno utilizzato una particolare tipologia di rete neurale, che viene chiamata graph neuronal network (rete neurale a grafo)- sottolinea Riccardo Zese, docente del Dipartimento di scienze chimiche, farmaceutiche ed agrarie dell’Università di Ferrara -. Questi graph neuronal networks) vanno a incapsulare quella che è una struttura grafica. Vanno un po’ a simulare quella che è la forma di questi composti. [..] Praticamente puoi vedere che gli atomi sono i nodi e i legami chimici sono gli archi di questo grafo.”

Questo tipo di rete neurale, oltre che in farmacologia, può essere utilizzata anche in altri campi, come lo studio dei social network: “I nodi – continua Zese – sono le persone e gli archi sono i collegamenti di queste persone, e quindi posso andare a vedere se una persona è collegata a un’altra persona, anche non direttamente”.
In maniera simile possono essere studiate le molecole in base alla loro struttura, valutando come le componenti fondamentali – gli atomi- sono collegati tra loro, e quali conformazioni possono avere maggiore efficacia come antibiotici.
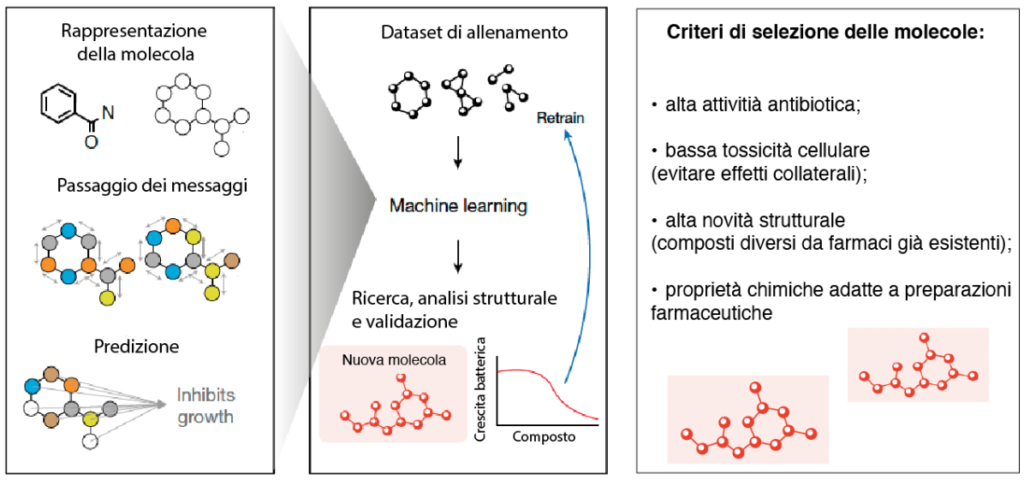
Il graph neural network è stato “allenato” su dataset generati in laboratorio, per poi predire la capacità di oltre 12 milioni di composti di inibire la crescita batterica e, al contempo, valutare la potenziale tossicità sulle cellule umane. Da un’ulteriore selezione e test sperimentali sono state individuate alcune molecole con attività antibiotica contro MRSA e un altro tipo di batterio, enterococco resistente alla vancomicina. Questi composti si sono dimostrati efficaci anche in modelli pre-clinici.
Capire cosa succede nella scatola nera rimuovendo uno a uno atomi e legami
Una novità cruciale dello studio, che è parte dell’ “antibiotic and AI project”, è che i ricercatori sono stati in grado di identificare il razionale utilizzato dal modello per identificare i composti con migliore attività antibatterica, a partire dalla struttura chimica.
È stato utilizzato un metodo di ricerca che, in pratica, rimuove uno ad uno gli atomi e i legami dei composti per valutare qual è il loro contributo al prediction score, cioè il punteggio assegnato in base all’attività antibiotica. Questa ricerca è stata condotta su 380 composti selezionati, poi testati sperimentalmente, e il razionale identificato mostra alcune caratteristiche strutturali conservate, associate all’attività antibiotica. Questo approccio ha permesso di identificare delle caratteristiche chiave per individuare nuove classi strutturali di antibiotici.
Ultimamente sono cresciuti gli sforzi per comprendere i criteri che guidano le scelte dei modelli di intelligenza artificiale, la cui difficile interpretazione è considerata un limite, come già riportato su Agenda17.
La possibilità di aprire la black box da parte ricercatori dell’MIT è legata alla tipologia di rete neurale utilizzata: essendo noti tutti i nodi di questa rete è possibile rintracciare il cosiddetto “passaggio di messaggi”, cioè il passaggio di informazioni che vengono utilizzate dal modello. “Riesco a vedere passo, passo quali sono stati i calcoli che sono stati fatti” spiega Zese. Lo stesso processo non è altrettanto facile da riprodurre in reti neurali più tradizionali, le cui black box rimangono, al momento, ancora oscure.

