“Negli ultimi anni, gli approcci computazionali sono diventati sufficientemente robusti e abbastanza sofisticati per influenzare significativamente il modo in cui identifichiamo i farmaci” afferma Jonathan M Stokes, professore associato alla McMaster University in Canada. Ne è un esempio il suo lavoro, recentemente pubblicato sulla rivista Nature chemical biology, in cui ha utilizzato strumenti di intelligenza artificiale per identificare un antibiotico contro il batterio multiresistente Acinetobacter diffuso in molti ospedali.
Più che un salto concettuale per ora c’è un aumento di potenza e molto dipende dai dati inseriti dall’uomo
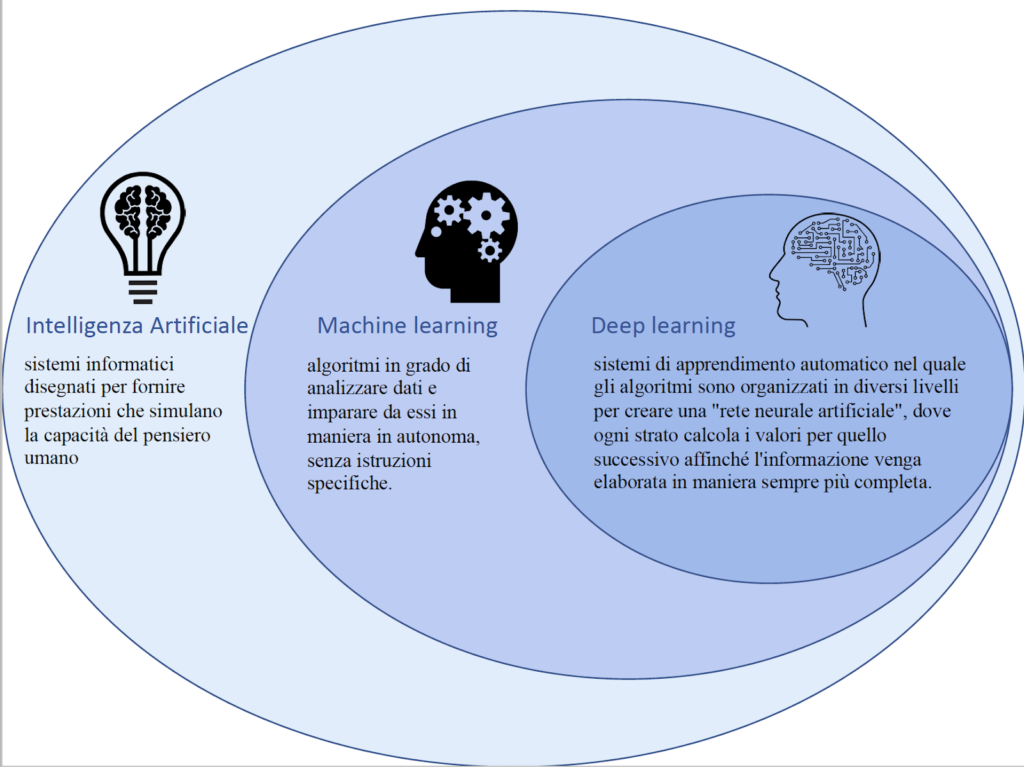
Sin dagli anni Ottanta , strumenti computazionali – che ora vanno sotto il cappello di “Intelligenza artificiale” (IA) – hanno accompagnato lo sviluppo della drug discovery, cioè la scoperta di nuovi farmaci. L’utilizzo di questi sistemi informatici è nato dalla necessità di gestire ed estrarre più informazioni possibili dai dati generati sperimentalmente.
“Quello che è cambiato dal 1990 ad oggi è fondamentalmente la potenza, però concettualmente stiamo parlando della stessa cosa- afferma Gabriele Costantino, direttore del Dipartimento di scienza degli alimenti e del farmaco dell’Università di Parma -. Il salto è stato più che altro tecnologico, ma non concettuale.”

Cosa intendiamo esattamente quando parliamo di IA? “Per Intelligenza artificiale si possono considerare moltissime cose. Diciamo che è il sovra insieme di tutto quello che permette alle macchine di prendere delle decisioni, e più o meno di avere dei comportamenti intelligenti – intelligenti da mettere tra virgolette, l’intelligenza è di chi le ha scritto quelle regole non tanto della macchina” afferma ad Agenda17 Riccardo Zese, ricecatore a tempo determinato del Dipartimento di scienze chimiche, farmaceutiche ed agrarie dell’Università di Ferrara. Una caratteristica di questi sistemi “è che cercano in qualche modo di agire in maniera autonoma” aggiunge.

L’agenzia americana che regolamenta la messa in commercio dei farmaci Food and drug administration (FDA), ha recentemente riportato un significativo aumento dell’utilizzo di strumenti di IA nello sviluppo di farmaci – nel 2021 ne sono stati presentati più di 100.
Strumenti di IA possono essere applicati in diverse fasi della drug discovery, dall’identificazione dei bersagli farmacologici al disegno di nuove molecole. Questo aspetto generativo ha le potenzialità di ampliare il cosiddetto “spazio chimico”, cioè l’insieme di tutte le molecole che esistono o possono essere sintetizzate.
Anche se ci sono alcuni esempi di farmaci disegnati de novo da IA, al momento questa capacità generativa non è ancora nel suo pieno potenziale secondo Costantino. “Forse il limite, più che di tecnologia, sta nella generazione di un dataset appropriato” commenta Zese.
I dati forniti in input alla macchina sono infatti un elemento cruciale nei sistemi di IA, per permetterle di imparare correttamente e fornire informazioni utili in uscita. Se questo non avviene, nel gergo computazionale si parla di “garbage in, garbage out” (spazzatura entra, spazzatura esce) spiega Stokes: dati di bassa qualità daranno risultati di bassa qualità.
Un nuovo approccio integrato può portare a breve a un cambio di paradigma
Secondo Costantino, gli strumenti di IA potrebbero consentire in futuro un cambiamento di paradigma nella drug discovery, permettendo di utilizzare un approccio sempre più integrato tra la ricerca di base e la clinica. “La capacità di interagire direttamente con intelligenza artificiale per raccogliere e integrare pezzi di informazione, che è molto complicato da fare senza”.
Le informazioni che circondano la vita di un farmaco sono molteplici: le caratteristiche della molecola per la sintesi, la disponibilità del principio attivo nelle diverse parti del mondo, il profilo tossicologico, la biodisponibilità – cioè la percentuale di farmaco in grado di raggiunge la circolazione sistemica- eventuali altri dati disponibili dalla clinica.
L’IA potrebbe aiutarci a connettere più velocemente tutte queste informazioni. “Un processo che in questo momento è sequenziale renderlo invece radiale” spiega Constantino “l’obiettivo idealmente dovrebbe essere di accorciare molto la fase, non soltanto di discovery, ma di development e poi di produzione industriale”.
L’antibiotico resistenza è un gravissimo pericolo che l’IA può affrontare
Un campo della drug discovery che secondo diversi ricercatori può trarre immediato beneficio dall’utilizzo di strumenti di IA è lo sviluppo di nuovi antibiotici. Questi farmaci in grado di combattere le infezioni batteriche hanno dato una svolta al trattamento e alla prevenzione delle malattie infettive, aprendo il capitolo della medicina moderna.
Per quale motivo abbiamo urgente bisogno di nuovi antibiotici? Perché sempre più batteri stanno sviluppando meccanismi di resistenza (Antimicrobial Resistance, AMR) ai farmaci già in commercio. Ricercatori e istituzioni sanitarie mondiali ci stanno ammonendo da tempo sui pericoli dell’“era post-antibiotica”: in mancanza di farmaci efficaci, non saremo più in grado di gestire le infezioni, riportando indietro il mondo di un centinaio di anni dal punto di vista sanitario.
Questo fenomeno avrà conseguenze su moltissime procedure mediche, che non saranno più sicure o possibili: dai trapianti d’organo, alla semplice estrazione di un dente.

Le cause dell’antibiotico-resistenza (AMR), già discusse su Agenda17, sono principalmente antropiche e legate all’utilizzo improprio di questi farmaci, sia in ambito umano che nei sistemi alimentari. Inoltre, l’aumento degli spostamenti ha favorito la diffusione dei ceppi resistenti. Da parte loro, i batteri sono in grado di trasferirsi vicendevolmente materiale genetico, distribuendo così anche i geni che conferiscono resistenza agli antibiotici.
Secondo l’ultimo rapporto dell’Osservatorio nazionale sull’impiego dei medicinali (OsMed) sull’uso degli antibiotici in Italia, nel 2021 prosegue il trend in riduzione dell’uso di antibiotici (-3,3% rispetto al 2020), ma i consumi si mantengono ancora superiori a quelli di molti Paesi europei. Inoltre, in Italia si fa un maggior ricorso ad antibiotici ad ampio spettro, cioè in grado di agire su diverse tipologie di batteri: una caratteristica utile nel caso in cui non sia nota la tipologia di patogeno si sta trattando, ma che purtroppo favorisce lo sviluppo di resistenze.
L’epidemia silenziosa: pochi sanno cos’è l’antibiotico resistenza
Secondo l’indagine “Gli italiani e gli antibiotici: informazione, utilizzo e consapevolezza del fenomeno dell’antibiotico resistenza” condotta dal CENSIS nel 2019, solo un italiano su due sa cos’è l’antibiotico-resistenza, e uno su cinque ne ha sentito parlare.
Anche oltreoceano, secondo Jonathan Stokes, professore associato alla McMaster University in Canada, l’AMR e le sue conseguenze non sono molto discusse al di fuori degli ambienti di ricerca “E questo è abbastanza preoccupante, considerando la dimensione del problema”.

Quali sono esattamente le dimensioni del problema? Una revisione inglese del 2014, ha provato a mettere dei numeri nero su bianco, riportando una previsione di 10 milioni di morti a causa dell’AMR nel 2050, una cifra equivalente alle morti per tumore oggi.
Parlando dei decessi già avvenuti, uno studio pubblicato l’anno scorso su Lancet ha stimando nell’anno 2019 quasi 5 milioni di morti associate ad AMR, di cui 1.3 milioni direttamente attribuibili all’AMR. Cifre simili ai decessi per Covid-19 nel primo anno della pandemia, che secondo le stime dell’Organizzazione mondiale della sanità (Oms) si aggirano attorno ai 3 milioni.
“Tutto quello che abbiam sentito per un paio d’anni quante persone stavano morendo per il Covid. Ma dell’antibiotico resistenza, che ha quasi gli stessi numeri, non se ne parla” considera Stokes. Sono due eventi che si sono sviluppati con tempistiche diametralmente opposte: la pandemia Covid-19, per quanto anch’essa prevista dagli scienziati, è diventato un evento di portata mondiale nel giro di pochi mesi. Invece “l’antibiotico resistenza è uno slow-moving problem– un problema che si è mosso lentamente” afferma Stokes “È iniziato in maniera tutto sommato gestibile, e poi i numeri sono cresciuti un po’, poi un pochino di più, fino a ritrovarci nello scenario in cui siamo ora”.
IA per lo sviluppo di nuovi antibiotici: un’impressionante accelerazione
Nel campo dell’ antibiotic-discovery, alcuni laboratori negli ultimi anni hanno iniziato a sfruttare strumenti di IA utilizzando diversi approcci “Guardano questo problema da angoli leggermente diversi. Ci sono alcuni gruppi che usano l’IA per la scoperta di peptidi antimicrobici. Ci sono un paio di gruppi che usano l’IA per modellare la diffusione dei geni della resistenza agli antibiotici dal punto di vista geografico. E poi ci sono un paio di gruppi, come noi, che fanno predizione delle proprietà molecolari con l’IA” spiega Stokes.
La maggior parte degli antibiotici utilizzati in clinica sono stati scoperti tra il 1940 e il 1960 attraverso lo screening dei metaboliti secondari prodotti dai microbi del suolo. Negli ultimi decenni, l’approccio allo sviluppo di nuovi antibiotici ha previsto principalmente la modifica dei farmaci già esistenti. Tuttavia, per i batteri multiresistenti questo approccio non è più applicabile, poiché occorrono antibiotici strutturalmente diversi.
Per trovare farmaci sostanzialmente diversi occorre fare lo screening di moltissime molecole “Ai livelli più alti possiamo studiare circa 2 milioni di molecole, e questo è molto costoso perché innanzitutto bisogna acquistarle, e viene impiegato un sacco di tempo per testarle sperimentalmente” spiega Stokes. “Mentre per fare il training di un modello computazionale bastano 10 mila molecole, due ordini di grandezza di meno”. E come tempistiche “possiamo fare circa 100 milioni di predizioni in circa 3 giorni. Per fare il test equivalente in laboratorio ci vorrebbero circa 10 anni”. La riduzione di tempi e costi di ricerca è quindi enorme secondo il ricercatore. A questo si aggiunge anche la possibilità di testare combinazioni sinergiche di farmaci.
Un caso eclatante: deep learning contro Iraquibacter
Stokes racconta ad Agenda17 che l’idea di applicare strumenti computazionali per lo sviluppo di nuovi antibiotici era nata anni prima al Massachusetts Institute of Technology (MIT) a Cambridge (USA), dalla collaborazione di microbiologi e bioinformatici che hanno deciso di unire le forze. L’obiettivo del primo studio era di trovare antimicrobici contro E.coli, il batterio più utilizzato nei laboratori. “Dopo aver completato questo progetto, ho iniziato a capire l’importanza di questi approcci computazionali per la generazione di nuovi antibiotici” spiega Stokes. “Se questo approccio funziona per E.coli, può funzionare anche per un patogeno clinicamente pericoloso?”.
Così il gruppo del ricercatore, che nel frattempo si è spostato in Canada, assieme a colleghi dell’MIT, si sono posti un obiettivo più ambizioso e clinicamente rilevante: Acinetobacter baumannii, chiamato anche “Iraqibacter”, il nemico invisibile emerso durante l’invasione americana in Iraq. Questo batterio multiresistente è spesso presente in ospedali e altri luoghi di assistenza sanitaria e può causare serie patologie come polmonite, meningite, infezioni del sangue e del tratto urinario.
Grazie a tecniche di deep-learning, i ricercatori sono riusciti a identificare una molecola, RS102895 ribattezzata abaucin, in grado di inibire la crescita di Acinetobacter in maniera selettiva, una caratteristica importante per evitare il successivo sviluppo di resistenze. Questi risultati, ottenuti su modelli animali, andranno ora validati a livello clinico.
Abaucin, era già nota per inibire il recettore per le chemochine CCR2, una molecola in grado di richiamare alcuni componenti del sistema immunitario, i monociti, dal midollo osseo a siti di infezione. Nei batteri invece ha dimostrato di avere un diverso meccanismo d’azione, bloccando il trasporto di alcuni componenti batterici, le lipoproteine, dalla membrana interna a quella esterna. Abaucin è quindi anche un esempio di come IA può spingere anche il “riposizionamento farmaceutico”, cioè trovare una nuova funzione per farmaci già esistenti, tagliando tempi e costi di produzione. (1.Continua)
(Articolo aggiornato al 7 luglio 2023)

